Parameters choosen
R-value: 95
Minimal size of secondary structure: 8
Minimal size of protein unit: 16
Maximal size of protein unit: 0
Pruning tree ? No
CI cut-off: 0.2
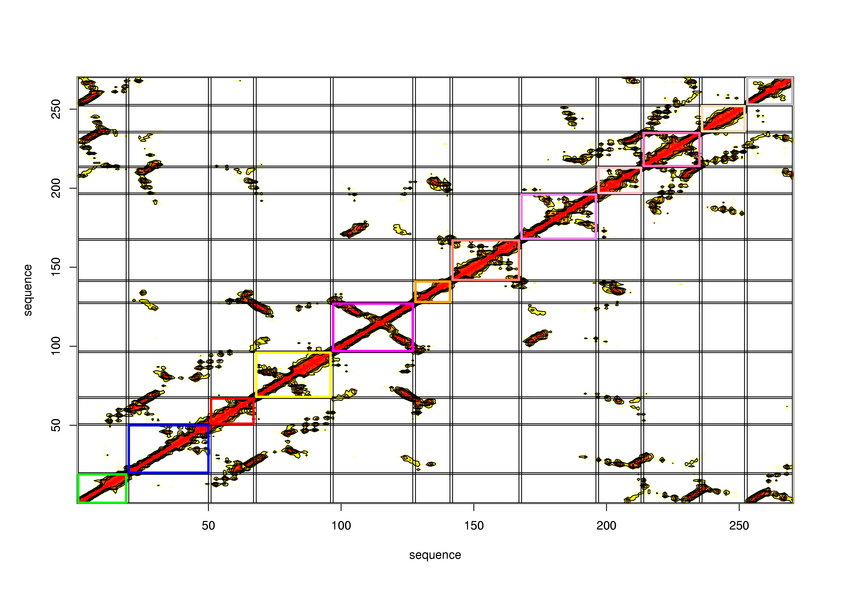
Cut-off distance between atom: 8
Delta value in logistic function: 1.5
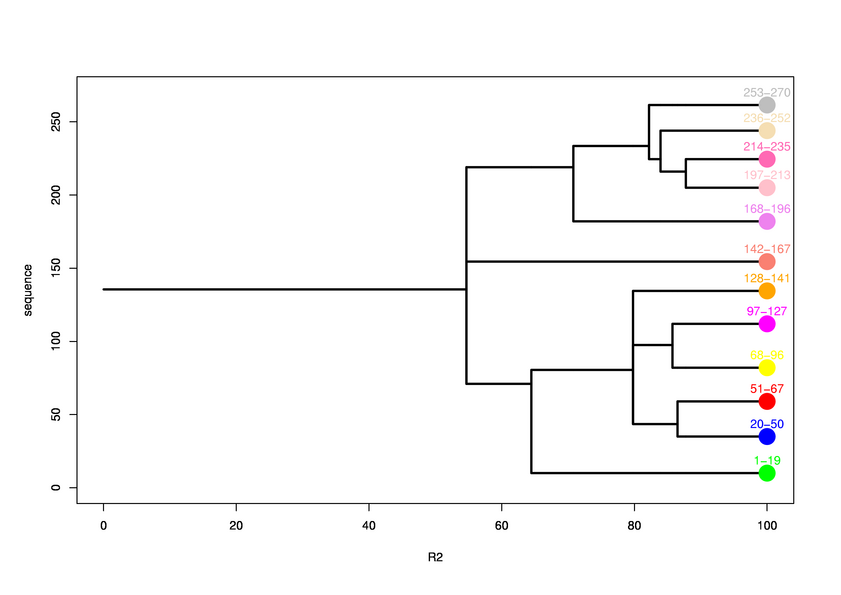
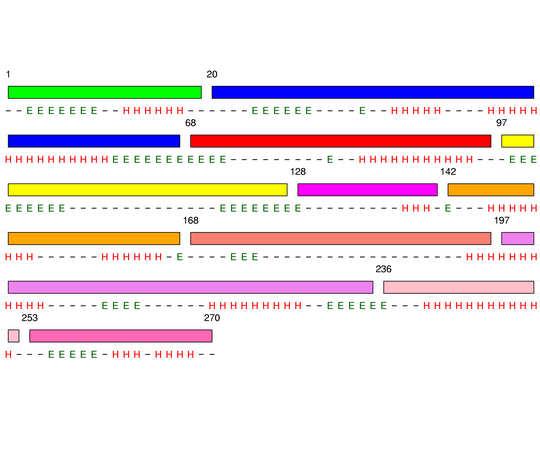
Hierarchical process of splitting
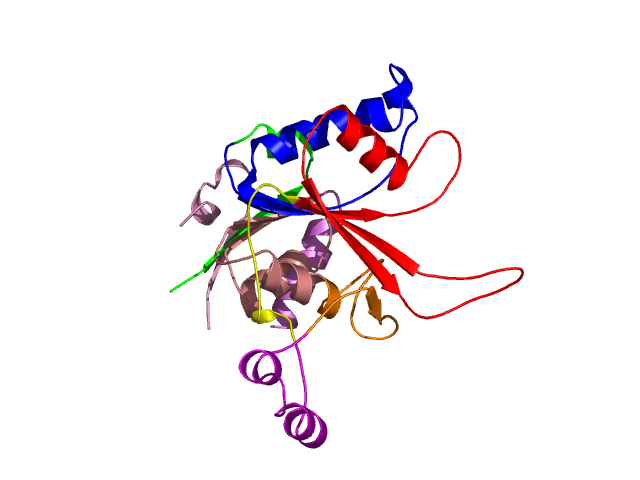
Visualisation of Protein units at all levels
Level 0

[1-141]:
CI=3.17
[142-167]:

CI=1.22
[168-270
]:
CI=2.65
Level 1
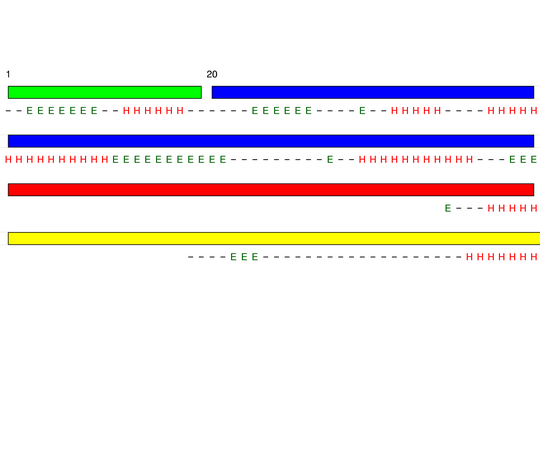

[1-19]:

CI=0.00
[20-141]:

CI=3.52
[142-167]:

CI=1.22
[168-270
]:
CI=2.65
Level 2
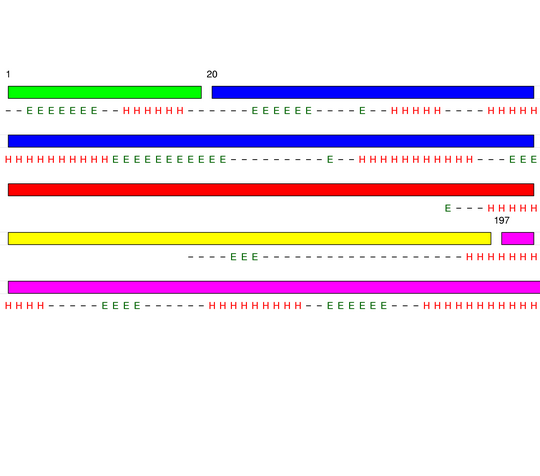
[1-19]:

CI=0.00
[20-141]:

CI=3.52
[142-167]:

CI=1.22
[168-196]:

CI=1.03
[197-270
]:
CI=2.62
Level 3
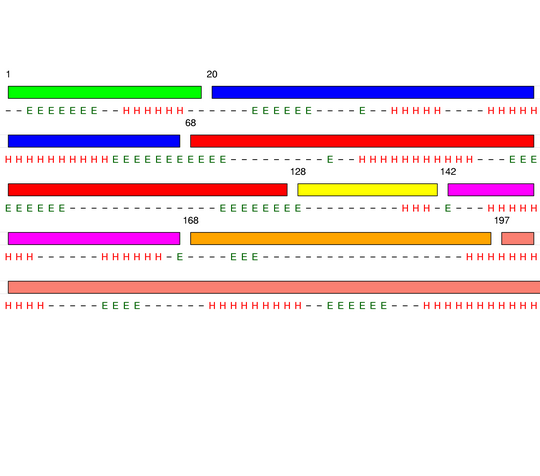
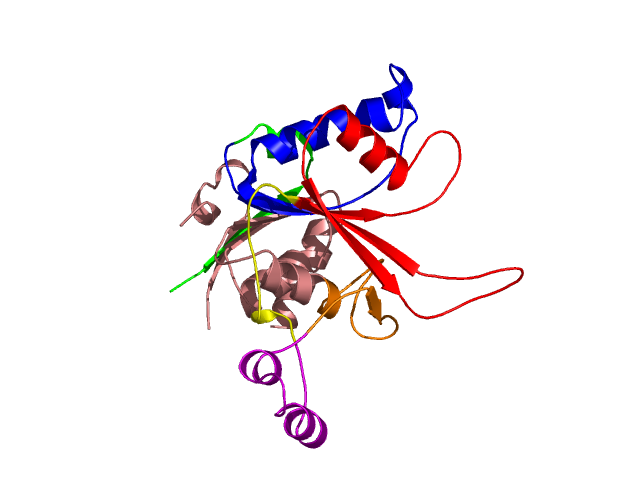
[1-19]:

CI=0.00
[20-67]:

CI=1.84
[68-127]:

CI=2.82
[128-141]:

CI=0.00
[142-167]:

CI=1.22
[168-196]:

CI=1.03
[197-270
]:
CI=2.62
Level 4
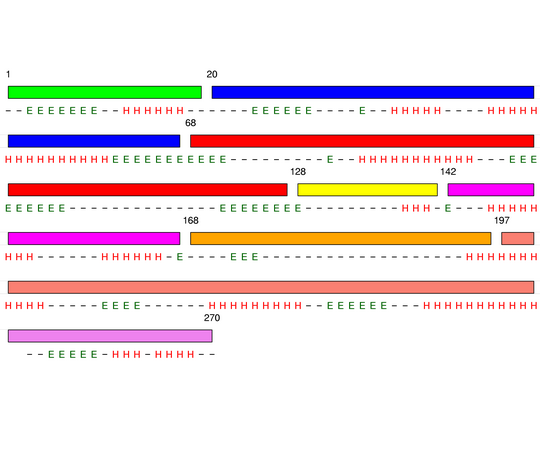
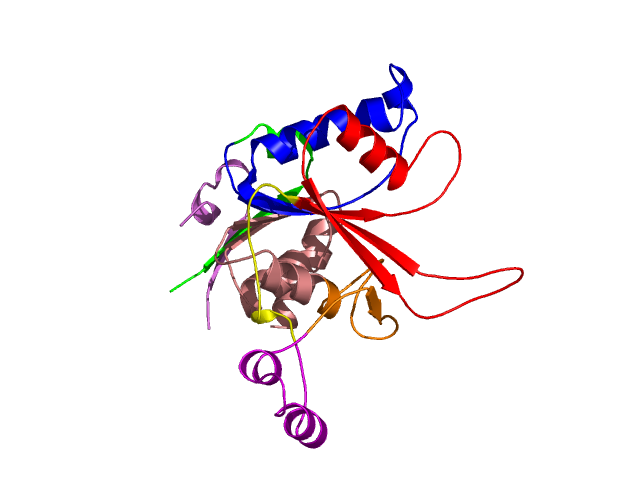
[1-19]:

CI=0.00
[20-67]:

CI=1.84
[68-127]:

CI=2.82
[128-141]:

CI=0.00
[142-167]:

CI=1.22
[168-196]:

CI=1.03
[197-252]:

CI=2.97
[253-270
]:
CI=0.00
Level 5
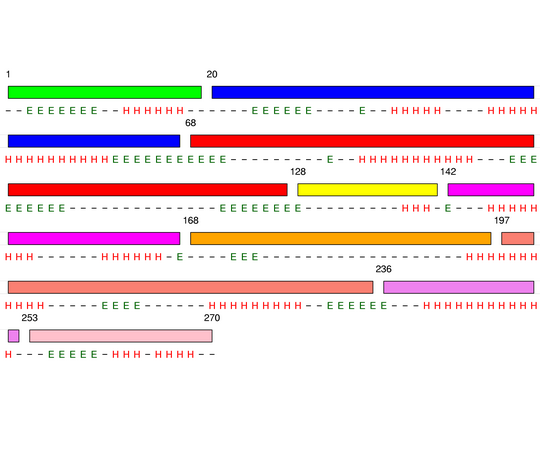
[1-19]:

CI=0.00
[20-67]:

CI=1.84
[68-127]:

CI=2.82
[128-141]:

CI=0.00
[142-167]:

CI=1.22
[168-196]:

CI=1.03
[197-235]:

CI=3.01
[236-252]:
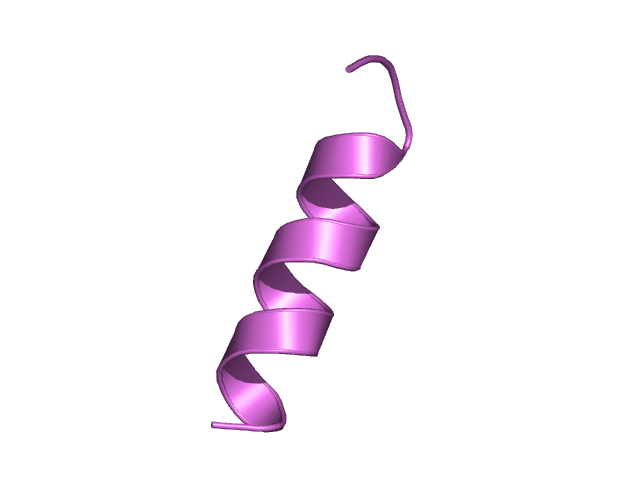
CI=0.00
[253-270
]:
CI=0.00
Level 6

[1-19]:

CI=0.00
[20-67]:

CI=1.84
[68-96]:

CI=1.04
[97-127]:

CI=2.64
[128-141]:

CI=0.00
[142-167]:
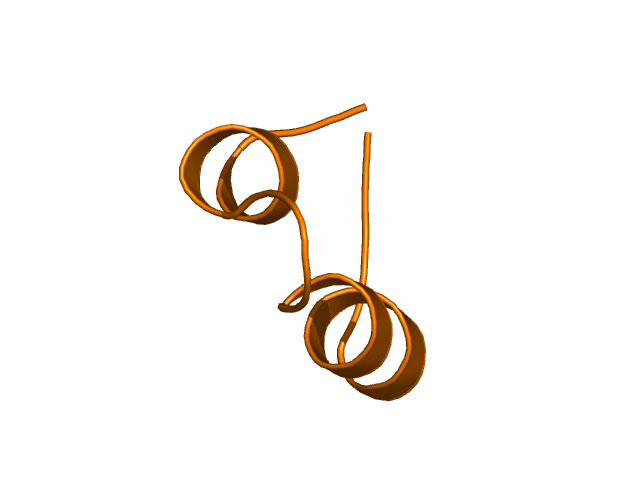
CI=1.22
[168-196]:

CI=1.03
[197-235]:

CI=3.01
[236-252]:
CI=0.00
[253-270
]:
CI=0.00
Level 7
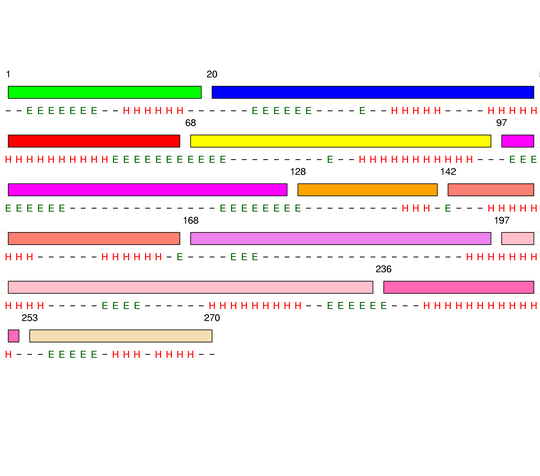
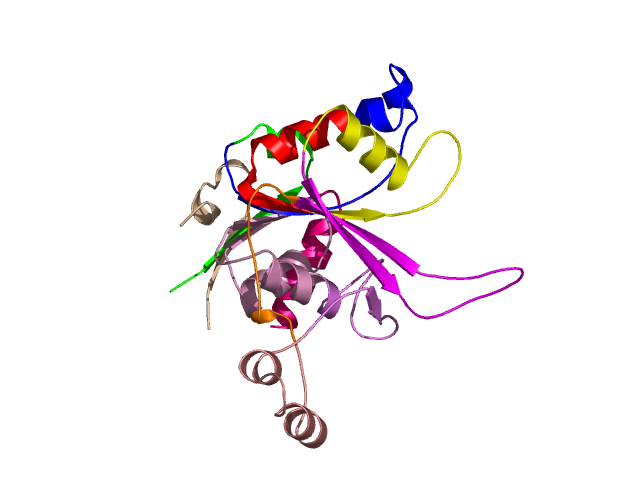
[1-19]:

CI=0.00
[20-50]:

CI=0.42
[51-67]:

CI=0.21
[68-96]:

CI=1.04
[97-127]:

CI=2.64
[128-141]:

CI=0.00
[142-167]:

CI=1.22
[168-196]:

CI=1.03
[197-235]:

CI=3.01
[236-252]:

CI=0.00
[253-270
]:
CI=0.00
Final level 8
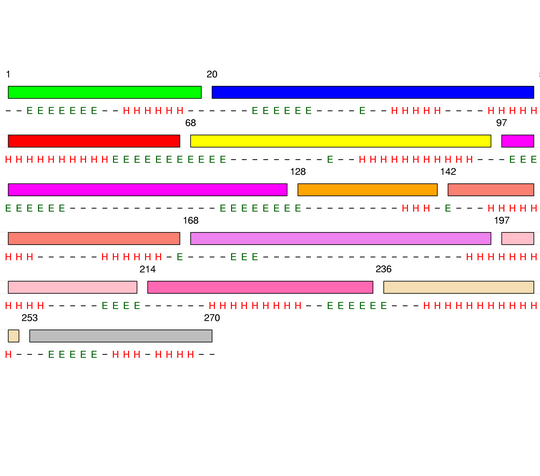
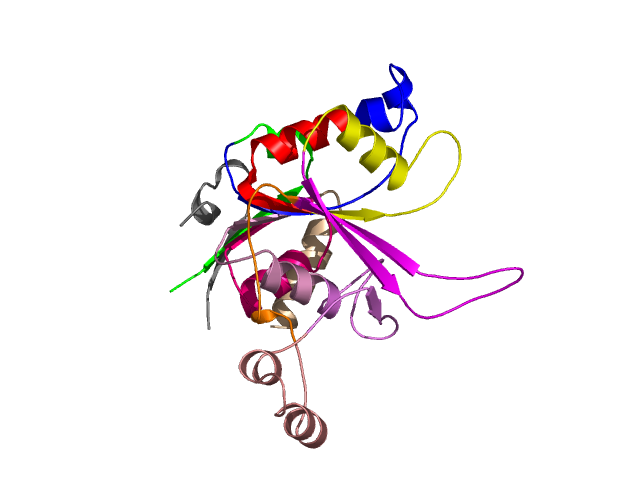
[1-19]:

CI=0.00
[20-50]:

CI=0.42
[51-67]:

CI=0.21
[68-96]:

CI=1.04
[97-127]:

CI=2.64
[128-141]:

CI=0.00
[142-167]:

CI=1.22
[168-196]:

CI=1.03
[197-213]:

CI=0.21
[214-235]:

CI=1.60
[236-252]:
CI=0.00
[253-270
]:
CI=0.00