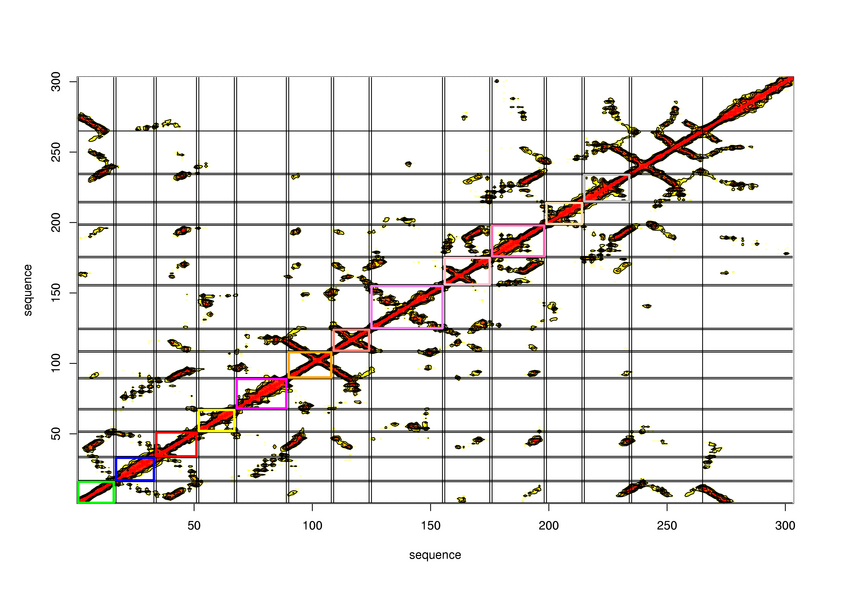
Parameters choosen
R-value: 95
Minimal size of secondary structure: 8
Minimal size of protein unit: 16
Maximal size of protein unit: 0
Pruning tree ? No
CI cut-off: 0.2
Cut-off distance between atom: 8
Delta value in logistic function: 1.5
Hierarchical process of splitting
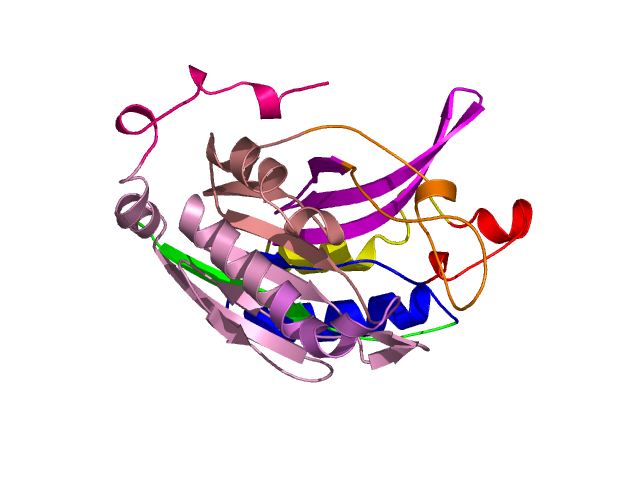
Visualisation of Protein units at all levels
Level 0
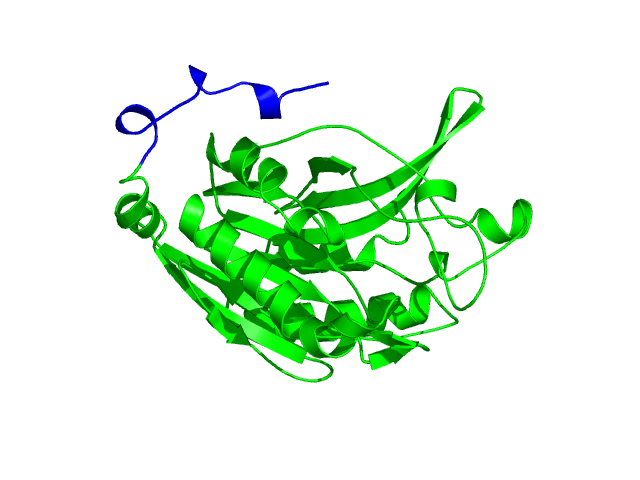
[1-286]:

CI=5.63
[287-303
]:
CI=0.00
Level 1
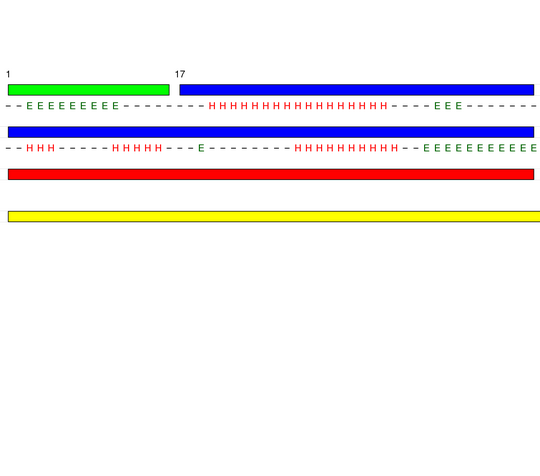
[1-16]:

CI=0.00
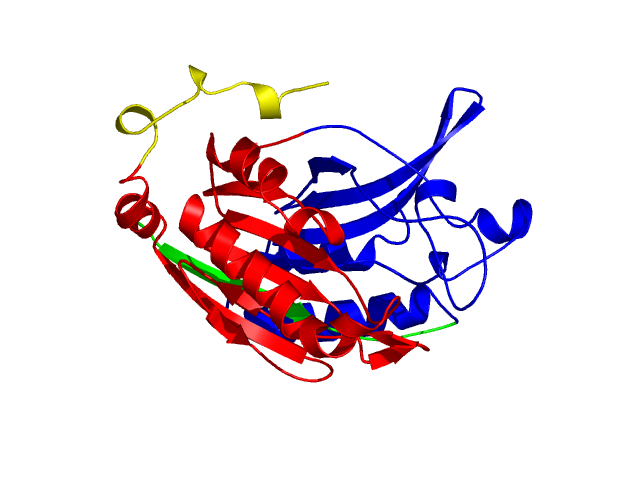
[17-155]:
CI=3.85
[156-286]:

CI=4.63
[287-303
]:
CI=0.00
Level 2

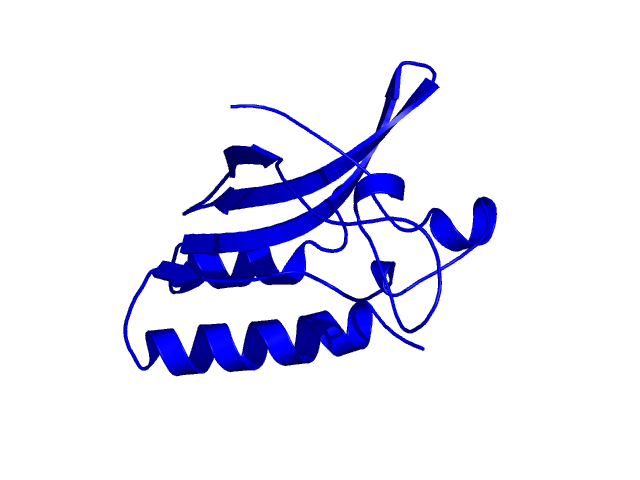
[1-16]:

CI=0.00
[17-155]:
CI=3.85
[156-198]:

CI=3.11
[199-214]:

CI=0.91
[215-286]:

CI=3.88
[287-303
]:
CI=0.00
Level 3

[1-16]:

CI=0.00
[17-124]:
CI=3.44
[125-155]:

CI=2.32
[156-198]:

CI=3.11
[199-214]:

CI=0.91
[215-286]:

CI=3.88
[287-303
]:
CI=0.00
Level 4

[1-16]:

CI=0.00
[17-51]:

CI=0.58
[52-67]:

CI=0.35
[68-124]:

CI=3.55
[125-155]:

CI=2.32
[156-198]:

CI=3.11
[199-214]:

CI=0.91
[215-286]:
CI=3.88
[287-303
]:
CI=0.00
Level 5
[1-16]:

CI=0.00
[17-51]:

CI=0.58
[52-67]:

CI=0.35
[68-89]:

CI=0.24
[90-124]:

CI=3.29
[125-155]:

CI=2.32
[156-198]:

CI=3.11
[199-214]:

CI=0.91
[215-286]:
CI=3.88
[287-303
]:
CI=0.00
Level 6
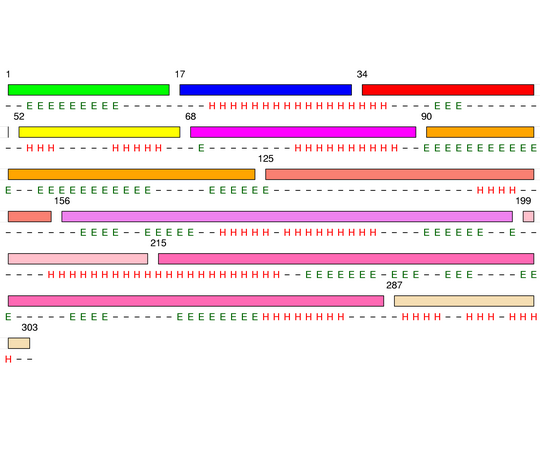
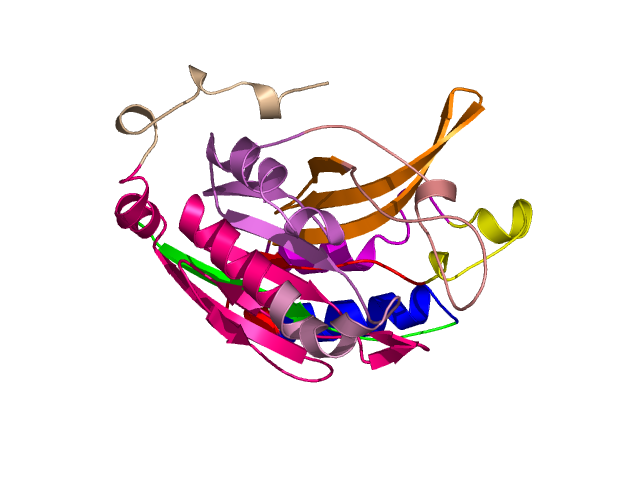
[1-16]:

CI=0.00
[17-33]:

CI=0.00
[34-51]:

CI=0.10
[52-67]:

CI=0.35
[68-89]:

CI=0.24
[90-124]:

CI=3.29
[125-155]:

CI=2.32
[156-198]:

CI=3.11
[199-214]:

CI=0.91
[215-286]:

CI=3.88
[287-303
]:
CI=0.00
Level 7
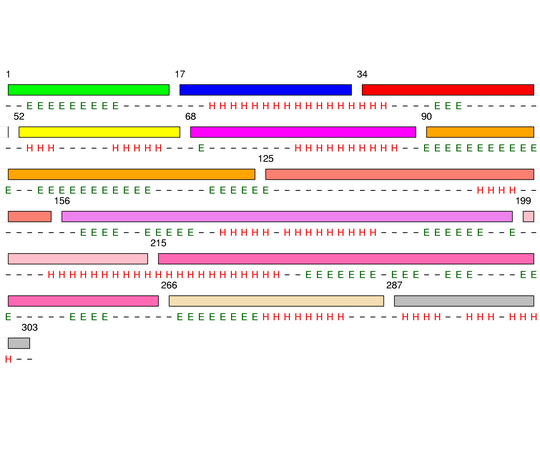
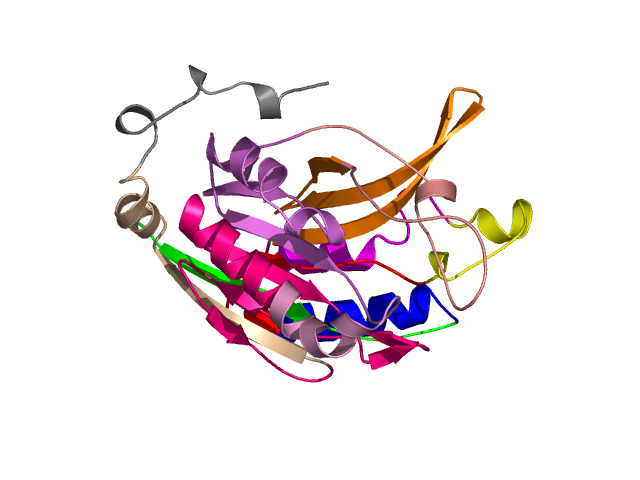
[1-16]:

CI=0.00
[17-33]:

CI=0.00
[34-51]:

CI=0.10
[52-67]:

CI=0.35
[68-89]:

CI=0.24
[90-124]:

CI=3.29
[125-155]:

CI=2.32
[156-198]:

CI=3.11
[199-214]:

CI=0.91
[215-265]:

CI=3.81
[266-286]:

CI=0.00
[287-303
]:
CI=0.00
Level 8
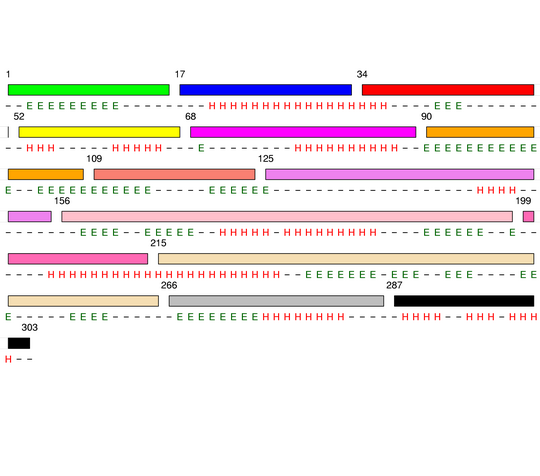
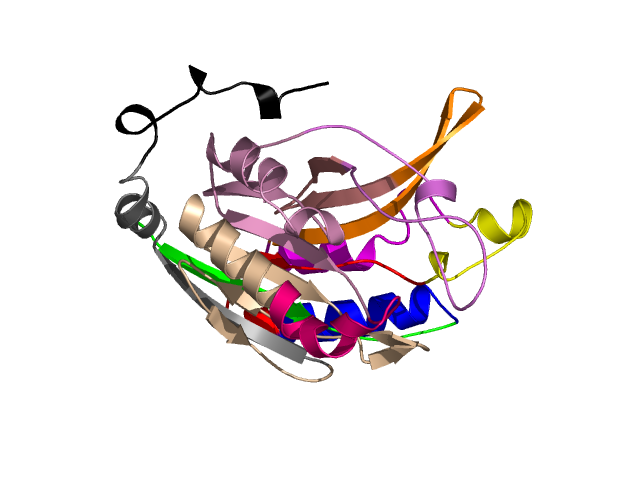
[1-16]:

CI=0.00
[17-33]:

CI=0.00
[34-51]:

CI=0.10
[52-67]:

CI=0.35
[68-89]:

CI=0.24
[90-108]:

CI=1.19
[109-124]:

CI=2.08
[125-155]:

CI=2.32
[156-198]:

CI=3.11
[199-214]:

CI=0.91
[215-265]:
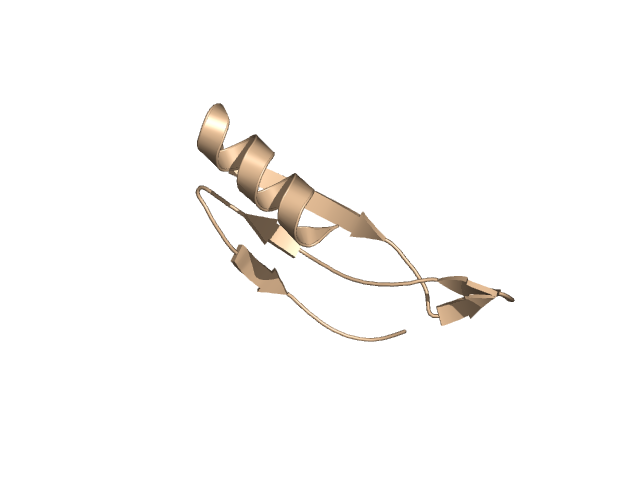
CI=3.81
[266-286]:

CI=0.00
[287-303
]:
CI=0.00
Level 9
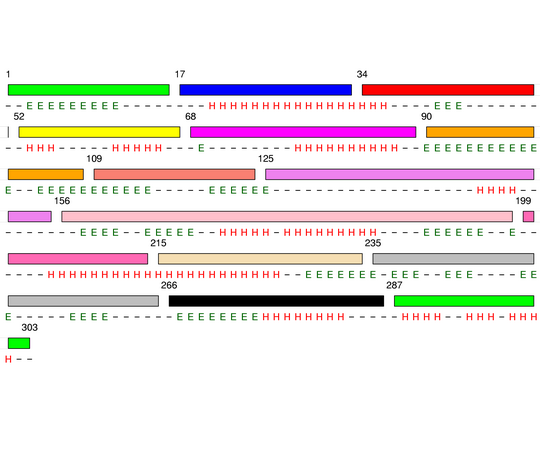
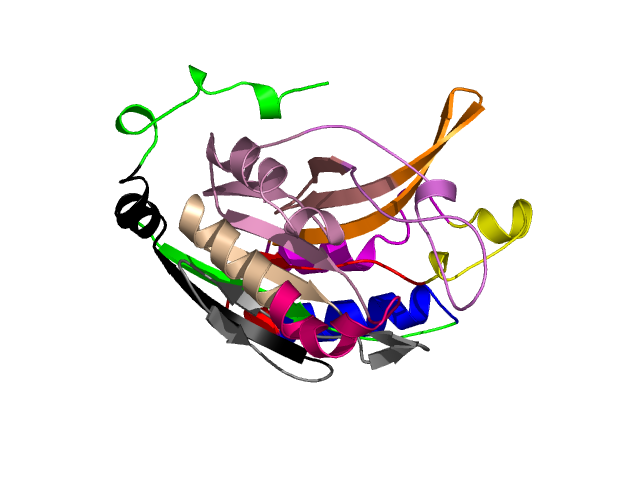
[1-16]:

CI=0.00
[17-33]:

CI=0.00
[34-51]:

CI=0.10
[52-67]:

CI=0.35
[68-89]:

CI=0.24
[90-108]:

CI=1.19
[109-124]:

CI=2.08
[125-155]:

CI=2.32
[156-198]:

CI=3.11
[199-214]:

CI=0.91
[215-234]:

CI=0.46
[235-265]:

CI=2.71
[266-286]:

CI=0.00
[287-303
]:
CI=0.00
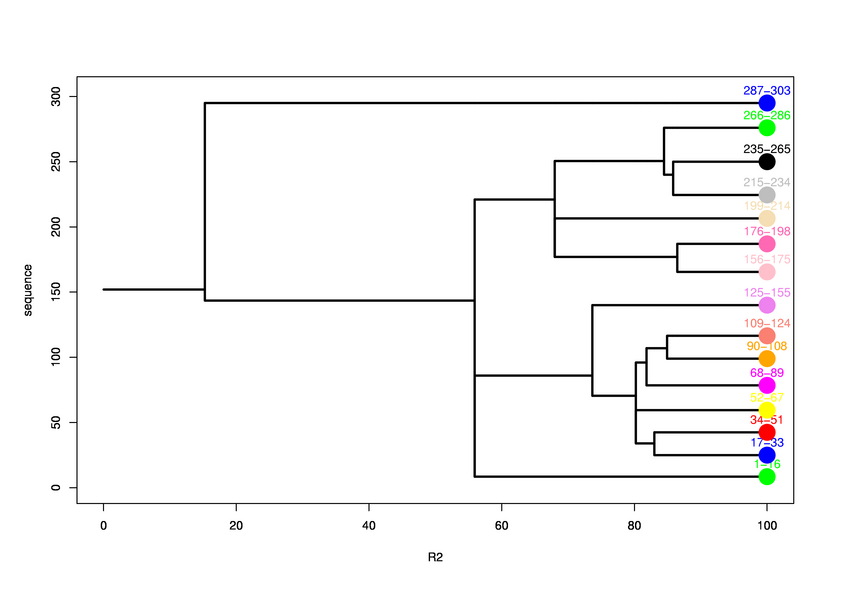
Final level 10
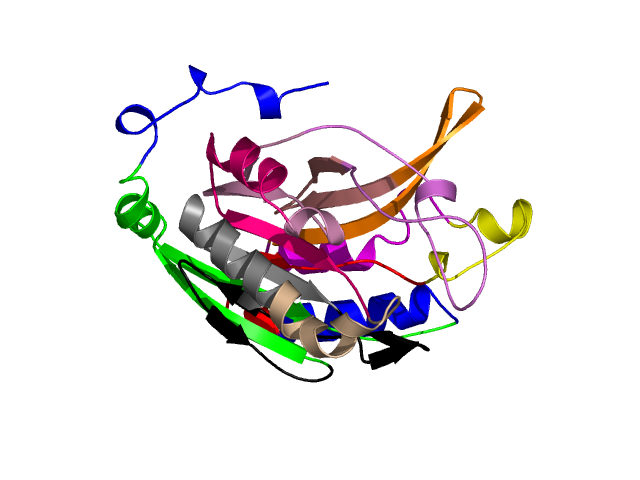
[1-16]:

CI=0.00
[17-33]:

CI=0.00
[34-51]:

CI=0.10
[52-67]:

CI=0.35
[68-89]:

CI=0.24
[90-108]:

CI=1.19
[109-124]:

CI=2.08
[125-155]:

CI=2.32
[156-175]:

CI=1.13
[176-198]:

CI=0.24
[199-214]:

CI=0.91
[215-234]:
CI=0.46
[235-265]:

CI=2.71
[266-286]:

CI=0.00
[287-303
]:
CI=0.00