Aurelie Bornot PhD
Year 2009
- Bornot A.
Analyse et prédiction de la relation séquence – structure locale et flexibilité au sein des protéines globulaires.
Doctorat de l'Université Paris Diderot - Paris 7 - Spécialité : Analyses de Génomes et Modélisation Moléculaire (2009) 250 p.
Résumé
La prédiction in silico de la structure tridimensionnelle d’une protéine à partir de sa séquence en acides aminés constitue un défi scientifique d’intérêt majeur. Il est à présent admis que les structures protéiques peuvent être décrites à partir d’un répertoire limité de structures locales récurrentes. Cette observation a conduit au développement de techniques de prédiction de la structure 3D par assemblage de fragments. Ces techniques sont aujourd’hui parmi les plus performantes. Dans ce contexte, la prédiction des structures locales constitue une première étape vers la prédiction de la structure 3D globale d’une protéine.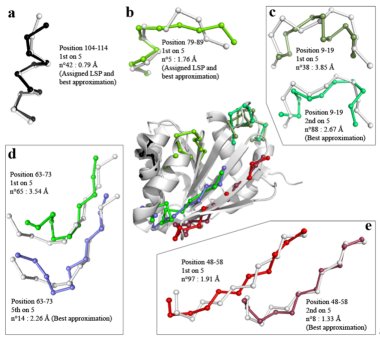 Mon travail de thèse porte principalement sur l’étude des structures protéiques locales à travers deux thèmes : (i) la prédiction des structures locales à partir de la séquence et (ii) l’analyse de la prédictibilité des structures locales en fonction de la flexibilité des structures protéiques. Ces études reposent sur une bibliothèque de 120 fragments chevauchants de 11 résidus de long précédemment développée au sein du laboratoire. Une méthode de prédiction des structures locales à partir de la séquence avait également été mise en place et permettait d’obtenir un taux de prédiction correct de 51 %. La prise en compte de données évolutionnaires couplée à l’utilisation de Machines à Vecteurs de Support a permis d’améliorer la prédiction des structures locales jusqu’à 63 % de prédiction correctes. De plus, un indice de confiance permettant d’évaluer directement la qualité de la prédiction et ainsi d’identifier les régions plus ardues à prédire a été mis au point. Par ailleurs, la structure des protéines n’est pas rigide. Ainsi, j’ai étendu notre analyse à l’étude la prédictibilité structurale des séquences d’acides aminés en fonction de leur flexibilité structurale au sein des protéines. Une analyse des propriétés dynamiques des structures locales a été menée en s’appuyant sur (i) les B-facteurs issus des expériences de cristallographie et (ii) les fluctuations du squelette polypeptidique observées lors de simulations de dynamique moléculaire. Ces analyses de la relation flexibilité-structure locale ont conduit au développement d’une stratégie de prédiction originale de la flexibilité à partir de la séquence. Nos différentes approches constituent une première étape vers la prédiction de la structure tridimensionnelle globale d’une protéine.
Mon travail de thèse porte principalement sur l’étude des structures protéiques locales à travers deux thèmes : (i) la prédiction des structures locales à partir de la séquence et (ii) l’analyse de la prédictibilité des structures locales en fonction de la flexibilité des structures protéiques. Ces études reposent sur une bibliothèque de 120 fragments chevauchants de 11 résidus de long précédemment développée au sein du laboratoire. Une méthode de prédiction des structures locales à partir de la séquence avait également été mise en place et permettait d’obtenir un taux de prédiction correct de 51 %. La prise en compte de données évolutionnaires couplée à l’utilisation de Machines à Vecteurs de Support a permis d’améliorer la prédiction des structures locales jusqu’à 63 % de prédiction correctes. De plus, un indice de confiance permettant d’évaluer directement la qualité de la prédiction et ainsi d’identifier les régions plus ardues à prédire a été mis au point. Par ailleurs, la structure des protéines n’est pas rigide. Ainsi, j’ai étendu notre analyse à l’étude la prédictibilité structurale des séquences d’acides aminés en fonction de leur flexibilité structurale au sein des protéines. Une analyse des propriétés dynamiques des structures locales a été menée en s’appuyant sur (i) les B-facteurs issus des expériences de cristallographie et (ii) les fluctuations du squelette polypeptidique observées lors de simulations de dynamique moléculaire. Ces analyses de la relation flexibilité-structure locale ont conduit au développement d’une stratégie de prédiction originale de la flexibilité à partir de la séquence. Nos différentes approches constituent une première étape vers la prédiction de la structure tridimensionnelle globale d’une protéine.
Summary
In silico prediction of protein structure from sequence is a major scientific challenge. It is now admitted that native 3D protein structures can be described by a limited set of recurring local structures. This observation led to the development of protein structure prediction techniques based on fragment assembly methods. Nowadays, these techniques are among the most effective. Protein local structure prediction is the first step toward the generation of global protein models.
This thesis work mainly focuses on two major questions: (i) protein local structure prediction from sequence and (ii) the analysis of local structure predictability according to protein structure flexibility features. These analyses were based on a library – previously developed in the laboratory – of 120 3D structural prototypes encompassing all known local protein structures. An associated local structure prediction method from sequence had also been created and yielded a correct prediction rate of 51 %. Here, we achieved a balanced improvement of the prediction rate by coupling evolutionary information with Support Vector Machines. A very satisfying correct prediction rate of 63 % was obtained. Moreover, for directly estimating the quality of the prediction, we developed a confidence index which enables to identify regions that are more difficult to predict. In the same way, protein structures are not rigid macromolecules. Hence, we also extended our analysis and addressed the question of the structural predictability of a sequence with regards to its structural flexibility properties inside protein structures. We analysed local structure flexibility features in proteins by relying on: (i) B-factors from X-ray experiments and (ii) backbone fluctuations observed in molecular dynamics simulations. Finally, an original flexibility prediction method from sequence was developed. Our different analyses are the first step toward the prediction of 3D global protein models.
Publications
- Bornot A., de Brevern A.G
Protein beta-turn assignments
Bioinformation (2006) 1(5) 153-5
-

 Doppelt O., Moriaud F., Bornot A., de Brevern A.G.
Doppelt O., Moriaud F., Bornot A., de Brevern A.G.
Functional annotation strategy for protein structures
Bioinformation (2007) Mar 19;1(9):357-9.
- Bornot A., Offmann B., de Brevern A.G.

 How flexible protein structures are ? New questions on the protein structure plasticity
How flexible protein structures are ? New questions on the protein structure plasticity
Bioforum Europe (2007) 11, 24-25
- Etchebest C., Benros C., Bornot A., Camproux A.-C., de Brevern A.G.

 A reduced amino acid alphabet for understanding and designing protein adaptation to mutation
A reduced amino acid alphabet for understanding and designing protein adaptation to mutation
European Biophysics Journal (2007) 36(8): 1059-69.
- Faure G., Bornot A., de Brevern A.G.

 Protein contacts, inter-residue interactions and side-chain modelling
Protein contacts, inter-residue interactions and side-chain modelling
Biochimie (2008) 90(4):626-39 - corrigendum 90(8):1264.
- Bornot A., Etchebest C., de Brevern A.G.

 A new prediction strategy for long local protein structures using an original description
A new prediction strategy for long local protein structures using an original description
Proteins (2009) 76(3):570-87.
- Ghozlane A., Joseph A.P., Bornot A., de Brevern A.G.

 Analysis of protein chameleon sequence characteristics
Analysis of protein chameleon sequence characteristics
Bioinformation (2009) 3(9), 367-369.
-

 Faure G.*, Bornot A.*, de Brevern A.G.
Faure G.*, Bornot A.*, de Brevern A.G.
Analysis of protein contacts into Protein Units
Biochimie (2009) 91(7):876-87.
-

 Tyagi M., Bornot A., Offmann B., de Brevern A.G.
Tyagi M., Bornot A., Offmann B., de Brevern A.G.
Analysis of loop boundaries using different local structure assignment methods
Protein Science (2009) 18(9):1869-81.
-

 Tyagi M., Bornot A., Offmann B., de Brevern A.G.
Tyagi M., Bornot A., Offmann B., de Brevern A.G.
Protein short loop prediction in terms of a structural alphabet
Computational Biology and Chemistry (2009) 33, 329-333
-

 Joseph A.P., Bornot A., de Brevern A.G.
Joseph A.P., Bornot A., de Brevern A.G.
Local Structure Alphabets
Protein Structure Prediction (2010)
Huzefa Rangwala and George Karypis eds., Wiley, 75-106.
-

 Pylouster J., Bornot A., Etchebest C., de Brevern A.G.
Pylouster J., Bornot A., Etchebest C., de Brevern A.G.
Influence of assignment on the prediction of transmembrane helices in protein structures
Amino Acids (2010) 39(5):1241-54.
-

 Joseph A.P., Agarwal G., Mahajan S., Gelly J.-C., Swapna L.S., Offmann B., Cadet F., Bornot A., Tyagi M., Valadié H., Schneider B., Etchebest C., Srinivasan N., de Brevern A.G.
Joseph A.P., Agarwal G., Mahajan S., Gelly J.-C., Swapna L.S., Offmann B., Cadet F., Bornot A., Tyagi M., Valadié H., Schneider B., Etchebest C., Srinivasan N., de Brevern A.G.
A short survey on Protein Blocks.
Biophysical Reviews (2010) 2(3):137-145.
-
 Bornot A., Etchebest C., de Brevern A.G.
Bornot A., Etchebest C., de Brevern A.G.
Predicting Protein Flexibility through the Prediction of Local Structures.
Proteins (2011)79(3):839-52.
*: both authors contributed equally to this work.
Alexandre G. de Brevern
Last Modification : March 2024
Last Modification : March 2024